Thème de recherche n°2 : Méthylation de l'appareil traductionnel
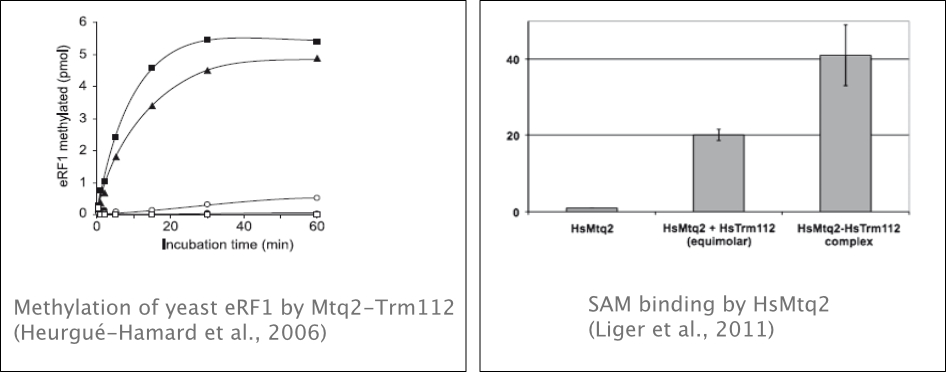
L’appareil traductionnel (ribosome, facteurs de traduction et ARNs de transfert) est la cible de nombreuses modifications post-transcriptionnelles et post-traductionnelles. La méthylation est une des modifications les plus courantes et de nombreuses protéines catalysent le transfert d’un groupement méthyle de la S-adénosylméthionine vers un substrat donné. Chez l’homme, un tiers de ces méthyltransferases (MTases) ont été liées au cancer ou à des maladies mentales. Cependant, leurs fonctions biologiques restent encore largement méconnues. Notre objectif est de comprendre les fonctions biologiques de la méthylation des ARN et des protéines impliquées dans la traduction. En effet, deux tiers des MTases connues ont pour substrat des composants de l’appareil traductionnel. Nous nous intéressons plus particulièrement à la protéine Trm112 de S. cerevisiae, qui active quatre méthyltransferases (MTases) ayant des substrats très différents, et qui se trouve à l’interface entre la synthèse du ribosome et sa fonction dans la traduction. Nous souhaitons élucider les fonctions de ces MTases en interaction avec Trm112. Nous avons montré que les facteurs de terminaison de la traduction de classe 1 sont méthylés chez les procaryotes (Escherichia coli, Chlamydia trachomatis), mais aussi dans le règne eucaryote (Saccharomyces cerevisiae, Homo sapiens, Mus musculus). Ces protéines sont essentielles à la libération d'une protéine fonctionnelle lorsque le ribosome rencontre un codon stop sur l’ARNm en cours de traduction. La méthylation se fait sur le résidu Gln du motif Gly-Gly-Gln (GGQ) conservé et impliqué dans la catalyse au centre de transfert peptidique du ribosome. Nous avons identifié les méthyltransférases impliquées dans ce processus, caractérisé leur fonction et leur structure (pour revue voir Graille et al., 2012). Grâce à des approches génétiques et biochimiques, nous avons caractérisé le mécanisme d'action de ces MTases et de la protéine activatrice Trm112 (Heurgué-Hamard et al., 2006, Liger et al., 2011).
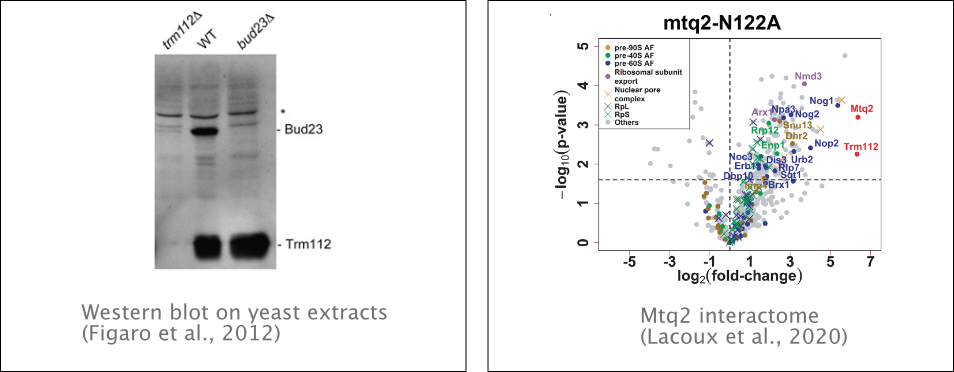
Trm112 interagit avec 4 MTases impliquées dans la modification de l’appareil de traduction: Mtq2 qui méthyle le facteur de terminaison eRF1, Trm11 et Trm9 qui méthylent des ARNt et Bud23 qui méthyle l’ARN ribosomique 18S. Nous avons montré que le complexe Bud23-Trm112 méthyle spécifiquement la base G1575 de l’ARNr 18S. En l’absence de Trm112, Bud23 est instable in vivo et ne peut plus interagir avec les pré-ribosomes naissants, avec pour conséquence un défaut de sous-unités 40S.
Plus récemment, nous avons montré que Trm112, via son interaction et stabilisation de Mtq2 a également un rôle dans la biogénèse de la grande sous-unité du ribosome. Mtq2 est en effet associée à différents précurseurs de 60S avec un rôle dans la production des ARNr matures et l’export de cette sous-unité. Mtq2 pourrait jouer un rôle de surveillance dans la formation de sites fonctionnels essentiels du ribosome (Lacoux et al., 2020).
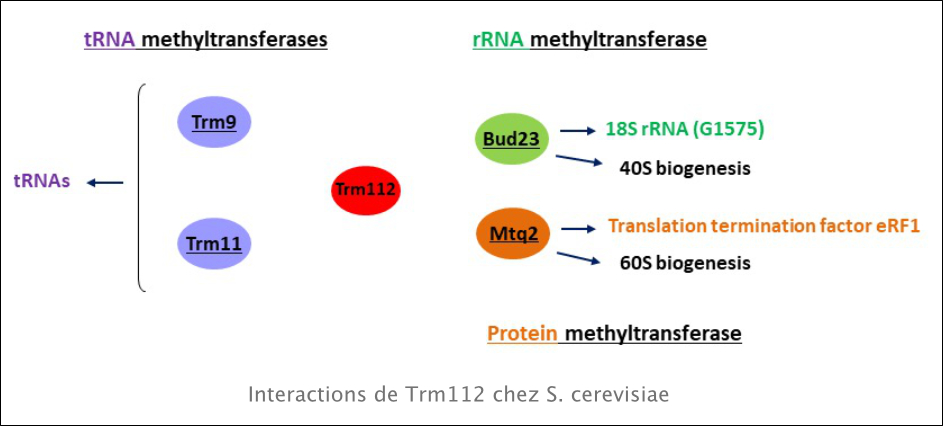
Trm112 est un activateur de MTases à une position stratégique, à l’interface entre la biogenèse du ribosome et sa fonction dans la traduction. Actuellement, nous cherchons à comprendre au niveau moléculaire le rôle de la méthylation dans la traduction, la biologie de la cellule en général, et le lien avec diverses pathologies. Nos expériences sont principalement réalisées dans la levure S. cerevisiae, modèle eucaryote très performant pour ses approches de génétique formelle et moléculaire. Nous utilisons les techniques courantes de biologie moléculaire (PCR, clonage, mutagenèse dirigée), des techniques de biochimie (purification de protéines à partir de systèmes d’expression homologues ou hétérologues, western blot, gradients de saccharose, immunoprécipitations) et des techniques de génétique de la levure.
Collaborateurs : Dr Marc Graille (Ecole Polytechnique, Palaiseau, France), Pr Denis L.J. Lafontaine (Université Libre de Bruxelles, Charleroi, Belgium), Dr Sarah Cianférani et Dr Christine Carapito (Institut Pluridisciplinaire Hubert Curien, Strasbourg, France)
